-
9. Scikit Learn을 활용한 train-test 데이터셋 나누기 (cross validation, K-fold)Data & ML & AI/Pandas & Scikit-Learn 2022. 8. 11. 01:08반응형
모델학습의 정확도, 과적합 여부를 확인하기 위해 데이터셋을 훈련용(train), 테스트용(test) 데이터셋으로 나누곤 합니다.
더 나아가서는 검증용(valid) 데이터셋으로 구분하기도 하죠.앞서 6.scikit-learn을 활용한 간단한 분류모델, 7.회귀모델에서도 잠깐 다루긴 했었지만, 아주 조금만 더 자세히 살펴볼까 합니다.
1. 데이터 가져오기
sklearn의 toy data 중 와인데이터를 사용해보겠습니다.
import pandas as pd from sklearn.datasets import load_wine wine = load_wine() X = wine['data'] Y = wine['target'] feature_names = wine['feature_names'] df = pd.DataFrame(X, columns = feature_names) print(wine['target_names']) print(Y) print(Y.shape) # ['class_0' 'class_1' 'class_2'] # [0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 # 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 # 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 # 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 # 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2] # (178,)target(Y)는 총 3개의 class로 이루어져 있습니다.
2. train-test 데이터셋 나누기
데이터셋 분할하기
train_test_split를 이용하면 간단하게 데이터셋을 분할할 수 있습니다.
물론 데이터 index 순서는 랜덤하게 섞여서 들어갑니다.- staratify : target 데이터(Y)의 분포 비율을 고려해서 적절히 train, test set 데이터를 만듭니다. 분류모델에서만 사용하는 것을 추천합니다.
- random_state : '777'이라는 고정된 랜덤시드를 사용합니다. 데이터셋을 나눌 때마다 데이터셋이 서로 다르게 만들어지는 것을 막습니다. 모델학습, 실험을 여러번 진행할 때, 데이터셋으로 인한 랜덤요소를 제거하고 각 모델 간 성능을 비교할 수 있습니다.
from sklearn.model_selection import train_test_split # X_train, X_test, y_train, y_test 의 순서에 유의해주세요 X_train, X_test, y_train, y_test = train_test_split(X, Y, stratify=Y, random_state=777) print(X_train.shape, y_train.shape) print(X_test.shape, y_test.shape) print(y_train) # (133, 13) (133,) # (45, 13) (45,) # array([1, 1, 1, 2, 1, 2, 2, 1, 0, 1, 1, 0, 2, 0, 1, 1, 1, 1, 0, 0, 2, 1, # 1, 0, 1, 0, 2, 0, 0, 2, 2, 0, 1, 2, 2, 1, 2, 2, 2, 2, 1, 2, 0, 0, # 0, 1, 0, 1, 1, 1, 0, 1, 1, 1, 2, 0, 1, 0, 2, 0, 2, 0, 0, 1, 0, 1, # 1, 2, 0, 2, 0, 2, 1, 0, 1, 1, 0, 0, 0, 1, 1, 2, 0, 0, 0, 0, 2, 1, # 1, 1, 0, 0, 2, 1, 0, 2, 0, 1, 2, 0, 2, 1, 1, 2, 0, 1, 1, 2, 2, 1, # 0, 1, 2, 2, 1, 1, 2, 0, 0, 0, 1, 1, 1, 1, 2, 0, 1, 1, 0, 2, 1, 0, # 2])모델 적용하기
정규화 과정 없이 대충 모델 돌리고 결과만 확인해보죠
from sklearn.linear_model import LogisticRegression model = LogisticRegression() model.fit(X_train, y_train) print("train set score :", model.score(X_train, y_train)) print("test set score :", model.score(X_test, y_test)) # train set score : 0.9699248120300752 # test set score : 0.95555555555555563. 랜덤성 없이 교차검증하기: Cross Validation (cross_val_score)
Cross Validation이란, train set을 한번에 다 사용하지 않고,
데이터셋을 일정 개수로 분할해 여러번 훈련하는 것을 의미합니다.전체 데이터셋을 (A,B,C) 3개로 나눴다면,
처음 훈련할 때는 A,B로만 학습 한 뒤 C로 테스트하고,
2번째에는 B,C로만 학습한 뒤 A로 테스트,
마지막 세번째에는 C,A로만 학습을 진행한 뒤 B로 테스트하는 방식입니다.이러면 모델 학습 과정에서의 과대/과소 적합 발생 가능성을 낮출 수 있습니다.
from sklearn.linear_model import LogisticRegression from sklearn.model_selection import cross_val_score model = LogisticRegression() scores = cross_val_score(model, X, Y, cv=3) # cv=3 --> cross validation 3번 print("cv set score :", scores, "\ncv mean score :", scores.mean()) # cv set score : [0.76666667 0.94915254 1. ] # cv mean score : 0.9052730696798493전체 평균점수는 0.90으로 그럴 수 있는데, 각 set의 개별 score가 많이 이상합니다.
이는, cross_val_score는 주어진 데이터를 순서대로 개수에 맞게 등분하기 때문입니다.
저희의 데이터처럼 class가 차례로 나열되어있는 분류모델학습용 데이터셋에는 치명적입니다.즉, 사진의 위쪽처럼 데이터를 적절하게 섞어주지 못했다는 것입니다.
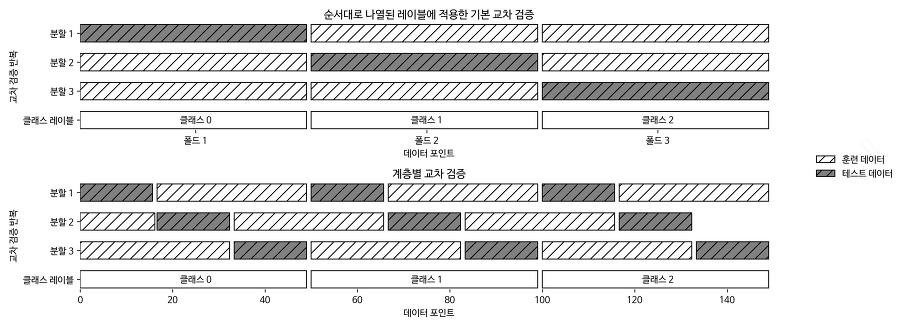
똑같이 생긴 사진이 여기저기 너무 많이 퍼져있어서 원본 출처가 뭔지를 모르겠습니다... 3. 랜덤성 있게 교차검증하기 : K-Fold(KFold)
교차검증에 랜덤성을 부여하는 방식은 여러가지가 있습니다.
cross_val_score를 하기 전에 train_test_split을 해도 되겠지만,
KFold를 사용하는 방법도 있습니다.from sklearn.linear_model import LogisticRegression from sklearn.model_selection import cross_val_score, KFold model = LogisticRegression() kfold = KFold(n_splits=3, shuffle=True, random_state=777) scores = cross_val_score(model, X, Y, cv=kfold) print("cv set score :", scores, "\ncv mean score :", scores.mean()) # cv set score : [0.98333333 0.93220339 0.93220339] # cv mean score : 0.94924670433145shuffle이 보다 골고루 되었음을 확인할 수 있습니다.
4. 임의분할 교차검증하기: ShuffleSplit

앞선 방법들은 모두 하나의 데이터는 단 한번만 사용되었습니다.
한번 사용된 데이터는 더이상 다른 fold에 속하지 않았다는 뜻입니다.반면, 데이터를 랜덤하게 선택하되, 뽑았던 데이터를 또 뽑는 방식으로 fold를 선택하는 방식도 있습니다.
중복으로 선택되는 데이터가 있다는 말은, 그만큼 한번도 선택되지 않는 경우도 발생한다는 뜻입니다.ShuffleSplit이 바로 그 역할을 수행합니다.
from sklearn.linear_model import LogisticRegression from sklearn.model_selection import cross_val_score, ShuffleSplit model = LogisticRegression() shuffle_split = ShuffleSplit(test_size=0.5, train_size=0.5, n_splits=3, random_state=777) scores = cross_val_score(model, X, Y, cv=shuffle_split) print("cv set score :", scores, "\ncv mean score :", scores.mean()) # cv set score : [0.97752809 0.95505618 0.93258427] # cv mean score : 0.9550561797752808반응형'Data & ML & AI > Pandas & Scikit-Learn' 카테고리의 다른 글
10. Grid Search: 머신러닝 모델 하이퍼파라미터 튜닝, 최적화 손쉽게 하기(feat. scikit learn) (0) 2023.03.30 [Pandas] pd.merge 속도 빠르게 바꾸기 (2) 2023.03.02 8. 판다스를 활용한 원핫인코딩(One hot encoding) (0) 2022.06.10 7. scikit-learn을 활용한 간단한 회귀모델(regression with LinearRegression) (1) 2022.06.08 6. scikit-learn을 활용한 간단한 분류모델 (classification with LogisticRegression) (0) 2022.06.03